library(tidyverse)Pipes
Introdução
A partir da versão 4.1.0, o R passou a oferecer o operador |> chamado de pipe (literalmente, cano)1. Este operador foi fortemente inspirado no operador homônimo %>% do popular pacote magrittr. Em atualizações mais recentes (versões 4.2.0 e 4.3.0) o operador ganho ainda mais capacidades.
O operador pipe carrega um objeto através de uma sequência de funções. A lista abaixo resume as principais propriedades do operador.
- Simplifica funções compostas. Na expressão
x |> f |> go operador|>aplica a funçãofsobre o objetoxusandoxcomo argumento def. Depois, aplica a funçãogsobre o resultado def(x). Isto é equivalente ag(f(x)). - Evita a definição de objetos intermediários. O uso de pipes evita que você precise “salvar” cada passo intermediário da aplicação de funções. Isto deixa seu espaço de trabalho mais limpo e também consome menos memória.
- Placeholder. Quando o objeto anterior não serve como o primeiro argumento da função subsequente, usa-se o placeholder para indicar onde ele deve ser inserido.
x |> f(y = 2, data = _). Isto funciona para selecionar objetos comomtcars |> _$mpg. - Função anônima. Em casos mais complexos, é necessário montar uma função anônima usando
x |> (\(y) {funcao})().
Aplicações comuns
tidyverse
O uso mais comum de pipes é junto com funções do tidyverse, que foram desenvolvidas com este intuito.
As funções do tidyverse (quase) sempre recebem um data.frame como primeiro argumento; isto facilita a construção de código usando pipe, pois basta encadear as funções em sequência.
filtered_df <- filter(mtcars, wt == 2)
grouped_df <- group_by(filtered_df, cyl)
tbl <- summarise(grouped_df, avg = mean(mpg), count = n())
mtcars |>
filter(wt > 2) |>
group_by(cyl) |>
summarise(avg = mean(mpg))A leitura do código fica mais “gramatical”: pegue o objeto mtcars filtre as linhas onde wt > 2 depois agrupe pela variável cyl e, por fim, tire uma média de mpg.
Pode-se terminar um pipe com uma chamada para um plot em ggplot2 para uma rápida visualização dos resultados.
mtcars |>
filter(wt > 2) |>
group_by(cyl) |>
summarise(avg = mean(mpg)) |>
ggplot(aes(x = as.factor(cyl), y = avg)) +
geom_col()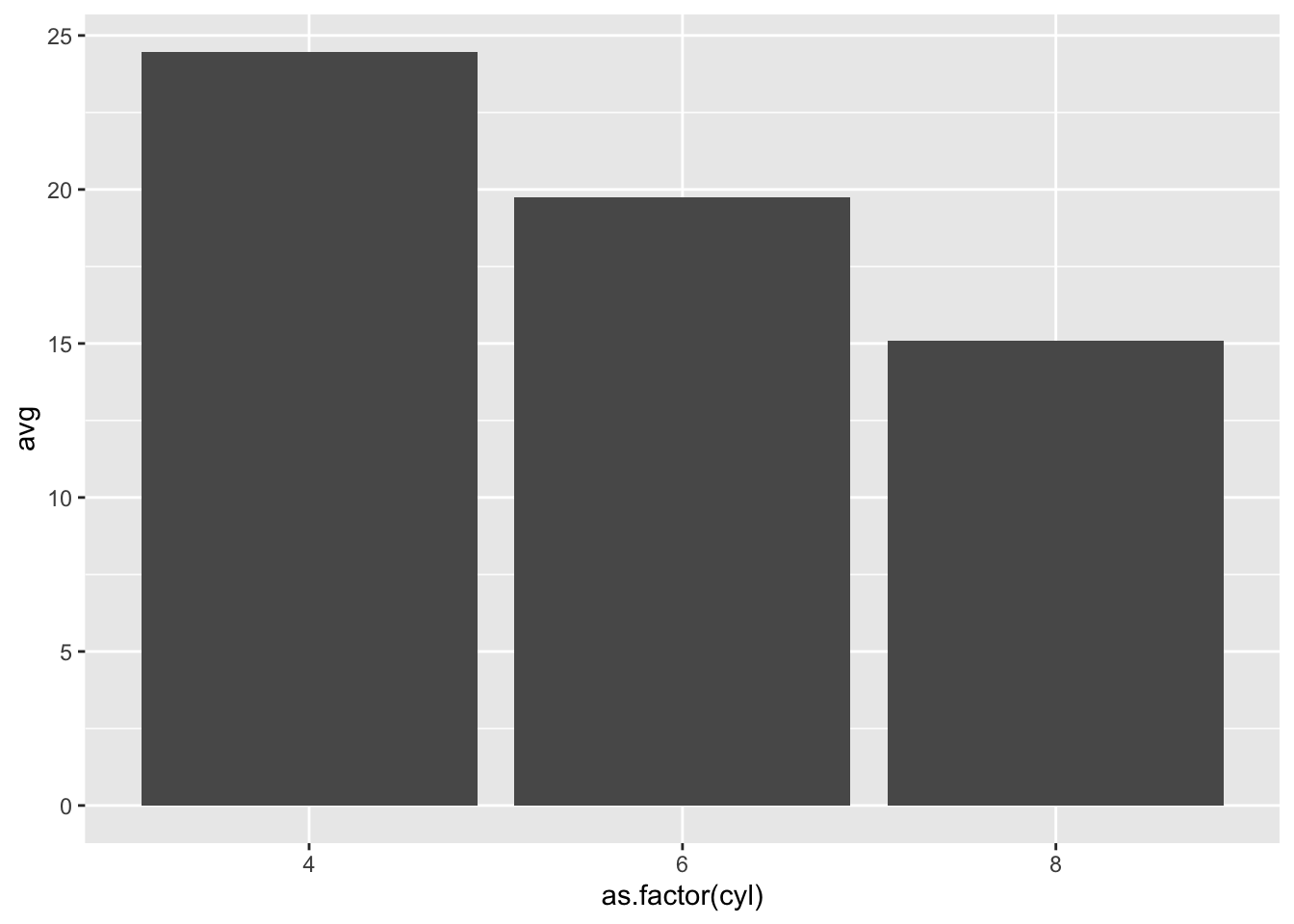
data.table
Na sintaxe do data.table é possível encadear as operação naturalmente usando [. Desde a atualização mais recente do R (>= 4.3.0) também é possível usar o pipe nativo
library(data.table)
dtmtcars <- copy(mtcars)
dtmtcars <- setDT(dtmtcars)
# Calcula a média de 'mpg' por 'cyl' e depois ordena segundo 'cyl'
dtmtcars[, .(mpg = mean(mpg)), by = cyl][order(cyl)] cyl mpg
<num> <num>
1: 4 26.66
2: 6 19.74
3: 8 15.10Pode-se quebrar o código acima usando o pipe nativo.
dtmtcars[, .(mpg = mean(mpg)), by = cyl] |>
_[order(cyl)] cyl mpg
<num> <num>
1: 4 26.66
2: 6 19.74
3: 8 15.10sf
O pacote sf também funciona bem com pipes pois há vários casos em que se quer aplicar múltiplas funções num mesmo objeto.
# Transforma um data.frame num objeto espacial (pontos)
# depois faz a interseção dos pontos num polígono e
# por fim limpa as geometrias
dat |>
st_as_sf(coords = c("lng", "lat"), crs = 4326) |>
st_join(poly) |>
filter(!is.na(gid)) |>
st_make_valid()O exemplo abaixo é emprestado do pacote censobr e mostra como combinar a manipulação de dados do dplyr com objetos espaciais manipulados via sf.
library(censobr)
library(geobr)
library(sf)
library(mapview)
# Importa alguns dados do Censo IBGE 2010
pop <- read_population(
year = 2010,
columns = c("code_weighting", "abbrev_state", "V0010")
)
# Calcula a população total das áreas de ponderação no Rio de Janeiro
df <- pop |>
filter(abbrev_state == "RJ") |>
group_by(code_weighting) |>
summarise(total_pop = sum(V0010)) |>
collect()
# Import o shape das áreas de ponderação do Censo
areas <- read_weighting_area(3304557, showProgress = FALSE)
areas |>
# Converte o CRS da geometria
st_transform(crs = 4326) |>
# "Limpa" as geometrias
st_make_valid() |>
# Junta com os dados do Censo
left_join(df, by = "code_weighting") |>
# Visualiza os dados num mapa interativo
mapview(zcol = "total_pop")Boas práticas
O guia de estilo do tidyverse propõe algumas orientações gerais sobre o uso de pipes. Abaixo eu reutilizao vários exemplos do guia.
Pipes longos
Primeiro, deve-se evitar de fazer um pipe long numa mesma linha. Esta recomendação visa melhorar a leitura do código. Também se recomenda deixar um espaço em branco antes do |>.
# Bom
iris |>
group_by(Species) |>
summarise(across(where(is.numeric), mean)) |>
ungroup() |>
pivot_longer(-Species, names_to = "measure") |>
arrange(value)
# Ruim
iris |> group_by(Species)|>
summarise(across(where(is.numeric), mean))|>ungroup() |>
pivot_longer(-Species, names_to = "measure") |> arrange(value)Funções longas
No caso de funções longas, deve-se quebrar/indentar o código.
#> Bom
iris |>
group_by(Species) |>
summarise(
Sepal.Length = mean(Sepal.Length),
Sepal.Width = mean(Sepal.Width),
Species = n_distinct(Species)
)
#> Bom
iris |>
group_by(Species) |>
summarise(Sepal.Length = mean(Sepal.Length),
Sepal.Width = mean(Sepal.Width),
Species = n_distinct(Species))
#> Ruim
iris |>
group_by(Species) |>
summarise(Sepal.Length = mean(Sepal.Length), Sepal.Width = mean(Sepal.Width), Species = n_distinct(Species))Pipes curtos
Pipes são úteis quando se aplica uma sequência de funções a um mesmo objeto; faz pouco sentido usar um pipe quando se aplica somente uma função.
#> Bom
plot(AirPassengers)
mean(AirPassengers)
#> Ruim
AirPassengers |> plot()
#> Ruim
AirPassengers |> mean(, na.rm = TRUE)
#> Bom: neste caso evite o pipe
plot(AirPassengers)
mean(AirPassengers, na.rm = TRUE)
#> Ruim
mtcars |> ggplot() + geom_point(aes(x = wt, y = mpg))
#> Bom: neste caso evite o pipe
ggplot(mtcars) +
geom_point(aes(x = wt, y = mpg))Pipes dentro de funções
É possível utilizar pipes dentro de funções para fazer pequenas transformações. Apesar disto, recomenda-se evitar isto. Colocar um pipe dentro de outro fluxo de código torna ele confuso; quase sempre vale a pena criar um objeto intermediário ao invés de usar o pipe.
O código abaixo, por exemplo, usa a base economics_long, que reúne um conjunto de séries de tempo, e indexa elas na primeira observação. Assim o primeiro valor de cada série tem valor igual a 100 e os valores subsequentes são “proporcionais” a este valor inicial.
Para chegar neste cálculo eu uso um pipe, dentro de uma sequência de pipes.
# Ruim: pipe dentro de pipe
economics_long |>
left_join(economics_long |>
filter(date == min(date)) |>
select(variable, base = value)
) |>
group_by(variable) |>
mutate(index = value / base * 100) |>
filter(date <= as.Date("1980-01-01"))A versão alternativa do código cria um objeto intermediário com os valores iniciais de cada uma das séries.
# Bom: cria objeto intermediário
base_index <- economics_long |>
filter(date == min(date)) |>
select(variable, base = value)
economics_index <- economics_long |>
left_join(base_index, by = "variable") |>
group_by(variable) |>
mutate(index = value / base * 100) |>
filter(date <= as.Date("1980-01-01"))Faço um gráfico do resultado final para tornar evidente o que está acontecendo.
ggplot(economics_index, aes(x = date, y = index)) +
geom_line() +
facet_wrap(vars(variable))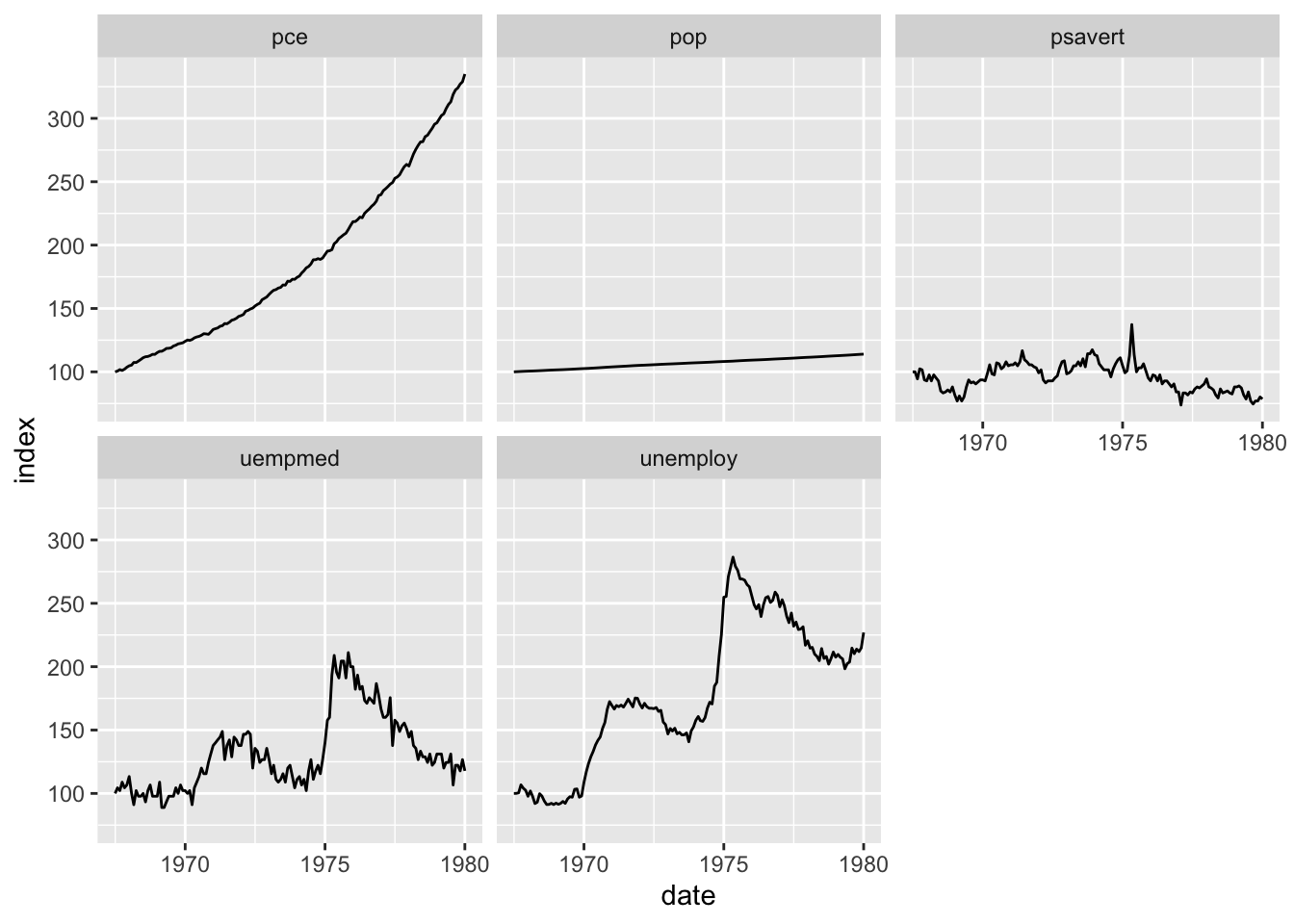
Resumo
O pipe é um operador simples que serve para deixar o código mais limpo. Sempre é uma boa escolha usar pipes quando se quer aplicar múltiplas funções num mesmo objeto. O custo do |> é mínimo em termos de eficiência; além disso, ele diminui a necessidade de criar objetos intermediários o que poupa memória do sistema.
O pipe nativo é embutido no R a partir da versão 4.1.0 enquanto o %>% exige library(magrittr). Vale notar que muitos pacotes carregam o %>% automaticamente. De fato, pacotes como dplyr, leaflet, mapview, rvest, gt, flextable e tantos outros são praticamente inutilizáveis sem pipes2.
Antes de pensar em usar o pipe nativo, vale a pena reforçar que ele foi criado recentemente. Isto significa que qualquer código ou pacote desenvolvido com uso de pipes vai exigir a versão 4.1.0 ou superior, o que pode exigir que o usuário atualize a sua versão do R. Além disso, algumas funcionalidades do pipe nativo mudaram nas versões 4.2 e 4.3 o que pode complicar ainda mais o uso do pipe. Em contrapartida, o pipe do magrittr é bastante estável e seu comportamento é consistente há muitos anos.
Qual pipe usar
Para a maioria dos casos, vale usar o pipe nativo do R, já que o %>% exige carregar um pacote adicional. Quando o placeholder for necessário, vale usar o %>% em vez do pipe nativo, devido à sua maior flexibilidade.
Referências
Footnotes
Para a lista completa de mudanças veja News and Notes.↩︎
Uma quantidade enorme de pacotes utiliza o
magrittrcomo dependência. Veja a página do CRAN.↩︎